eBot6User
Inhaltsverzeichnis
Voraussetzungen
Für eBot benötigen Sie:
- Einen Mac (MacOS X 10.5 oder höher) oder PC (Windows XP SP2 oder höher)
- Java Runtime Environment (JRE) Version 1.6 oder höher
Starten von eBot
Der Link zu eBot ist über die Hauptseite eBot 6 auf der Unterrichtshomepage von Matthias Baltisberger verfügbar.
Wenn Sie eBot nicht über diesen Link starten können, gehen Sie auf die Seite Troubleshooting und folgen Sie den Anweisungen.
Wenn eBot startet, erscheint ein Dialog mit Hinweisen zum Copyright und Angaben zur Nutzung und zu eventuellen Kosten für diese Applikation. Beim ersten Mal müssen Sie diese Hinweise lesen und akzeptieren.
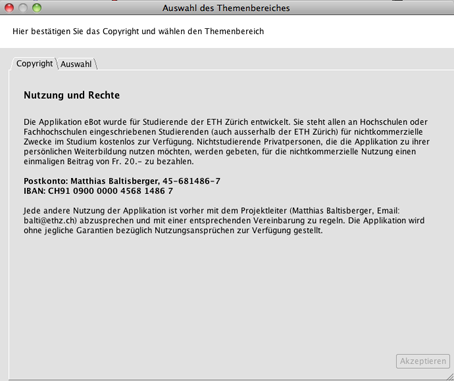
Sobald Sie das Copyright akzeptiert haben, wird der Reiter Auswahl aktiviert. Klicken Sie auf Auswahl, um die Startoptionen sichtbar zu machen.

Als Optionen stehen die beiden Bereiche Systematik und Dendrologie zur Verfügung. Nach Ihrer Wahl klicken Sie auf OK, dann startet eBot.
Gliederung des Fensters von eBot 6
Viele Dinge und Funktionen sind selbsterklärend und werden deshalb hier nicht vorgestellt.
Das Fenster von eBot 6 besteht aus 4 Komponenten:
1) Menübalken und Toolbars enthalten die allgemeinen Einstellungen:
Menübalken mit "Programm", "Moduswahl" (z.Z. nur mit der Standardeinstellung "Lernen") und "eBot".
Toolbars:
- Liste: Insgesamt stehen in eBot 6 632 Taxa auf dem Level Art zur Verfügung: 1 Pilz, 4 Flechten, 5 Moose, 19 Farne, 12 Gymnospermen und 591 Angiospermen. Über die Auswahl einer Liste kann der Stoffumfang eingeschränkt werden.
- Liste "200 CH" enthält jene 200 Arten, die von der Zertifizierungskommission für das Zertifikat 200 festgelegt wurden.
- Liste "200 Leuchtmann" enthält 200 Arten; diese sind Prüfungsstoff (Teil Artenkenntnis) für Studierende der Biologie, Pharmazie und Umweltnaturwissenschaften an der ETH Zürich (4. Semester, Prof. Dr. Adrian Leuchtmann).
- Die Liste "200 ZH" ist die Zürcher Liste für das Zertifikat Stufe 200; diese Liste "200 ZH" unterscheidet sich von der Liste "200 CH" in 29 Arten.
- Weitere Listen enthalten die für die Zertifizierungsstufen verlangten Arten: Liste "400" die Arten für das Zertifikat Stufe 400 resp. Liste "600" die Arten für das Zertifikat Stufe 600 (Arten für das Zertifikat Stufe 200 siehe Listen "200 CH" und "200 ZH"). Die Listen "400–200 ZH", "600–200 ZH" und "600–400" dienen dem stufenweisen Lernen (Einschränkung auf die bei den Stufen hinzukommenden Arten).
- Liste "600 Zeiger" umfasst jene Arten der Liste "600", die für die Feuchtezahl (F), die Reaktionszahl (R, pH-Wert des Bodens) und die Nährstoffzahl (N) extreme Zeigerwerte aufweisen. F1: sehr trocken, F1.5: trocken, F4.5: nass, F5: überschwemmt bzw. unter Wasser; R1: stark sauer, R5: basisch; N1: sehr nährstoffarm, N5: sehr nährstoffreich bis überdüngt. Bitte beachten Sie: Arten mit Zeigerwerten 2 oder 4 können ebenfalls gute Zeigerarten sein; diese Arten werden in eBot als „mässige Zeiger“ bezeichnet, sie sind aber nicht in der Liste "600 Zeiger" enthalten.
- Liste "Alle Taxa" ist die vollständige Liste mit allen 632 Taxa in eBot.
- Liste "PHARMBIO" enthält 79 Arten; diese sind Prüfungsstoff in der Veranstaltung „Pharmazeutische Biologie“ (Prof. Dr. Karl–Heinz Altmann).
- Reihenfolge: "taxonomisch" entspricht der Reihenfolge im phylogenetischen Baum, "zufällig" präsentiert die Taxa in nicht geordneter Reihenfolge.
- Modus: Klick auf "Modus: Lernen" wechselt auf "Modus: Abfragen" und umgekehrt.
- 5 Schaltflächen für das Erstellen und Bearbeiten eigener Listen.
- Straffen: Klick auf "Straffen" schliesst alle Knoten im phylogenetischen Baum ausser den Knoten, die zum angewählten Taxon führen.
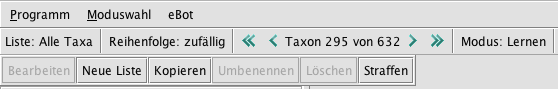
Toolbars mit den Einstellungen "Liste: Alle Taxa", "Reihenfolge: zufällig", "Modus: Lernen", sichtbar ist Taxon 295 (von 632).

Toolbars mit den Einstellungen "Liste: 200 Leuchtmann", "Reihenfolge: taxonomisch", "Modus: Abfragen", sichtbar ist Taxon 5 (von 200); rechts die Felder der gefragten Taxa zum Eintippen der Namen.
2) In der Mitte ist der Bildteil mit den 4 Reitern "Herbar", "Portrait", "Detail" und "Standort" für die Auswahl der entsprechenden Bilder.
3) Rechts ist der Informationsteil mit den 4 Reitern "Morphologie", "Ökologie", "Interessantes" und "Namen".
- Morphologie zeigt die Merkmale des ausgewählten Taxons sowie die Merkmale von übergeordneten Taxa.
- Ökologie enthält die Angaben zu Standort, Höhenverbreitung und ev. Zeigereigenschaften.
- Unter Interessantes finden Sie Angaben zur Verwendung (z.B. technische Nutzung, Nahrungsmittel, pharmazeutische Verwendung) und zur Giftigkeit.
- Namen enthält Synonyme und z.T. deutsche Namen.
4) Links befindet sich ein phylogenetischer Baum, dort sehen Sie die Position der Taxa im phylogenetischen System. Über diesen Baum lässt sich der Stoff und damit auch das Lernen beeinflussen:
- Klick auf "+" resp. "-" öffnet resp. schliesst den Knoten; Klick auf "Straffen" im Toolbar schliesst alle Knoten im Baum ausser den Knoten, die zum angewählten Taxon führen.
- Klick auf das farbige Icon definiert die hierarchische Stufe, auf der gearbeitet wird (genannt Level); im Bild ist die Stufe "Art" ausgewählt, ersichtlich daran, dass die Artnamen schwarz und die Namen auf den anderen Stufen grau sind.
- Klick auf den Kreis wählt dieses Taxon als oberstes Taxon des Arbeitsbereiches aus (genannt Scope); im Bild ist die Standardeinstellung "Alle Taxa" angewählt. Dies entspricht der Auswahl aller Taxa der aktuell ausgewählten Liste.
Beispiel: Ein Klick auf den Kreis neben "Amaryllidaceae" schränkt den Arbeitsbereich ein auf die Arten in Amaryllidaceae, die in der aktuell ausgewählten Liste vorkommen. - Klick auf einen (schwarzen) Namen wählt dieses Taxon aus (genannt Focus); im Bild ausgewählt ist "Allium ursinum" (blau hinterlegt); die zum ausgewählten Taxon gehörenden höheren Taxa sind grün geschrieben.
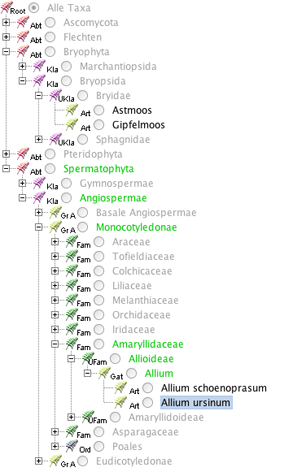
Einstellungen im gezeigten Baum: Level "Art", Scope "Alle Taxa", Focus "Allium ursinum".
Arbeiten im "Modus: Abfragen"
In der Abfrage kann das Erkennen von Arten sowie das Wissen über ihre Systematik (höhere Taxa) getestet werden. Die Namen der aktiven Taxa sind anonymisiert, im phylogenetischen Baum (links) steht statt eines gefragten Namens "Welches Taxon ist das?", im Informationsteil (rechts) sind die Namen durch die hierarchische Stufe ersetzt. Beim Abfragen muss der entsprechende wissenschaftliche Name in das richtige Feld eingetragen und mit Enter bestätigt werden. Bei richtiger Antwort wird das Namensfeld grau (im Bild "Populus tremula") und der Kursor springt ins nächste Feld, bei falschen Antworten (im Bild "Populaceae" ist falsch, es ist die Familie "Salicaceae") erfolgt ein entsprechender Hinweis (siehe Bild). Der Name muss nicht nur richtig sein, sondern auch richtig geschrieben werden; Endungsfehler in den Namen werden kommentiert. Synonyme und deutsche Namen werden ebenfalls kommentiert.
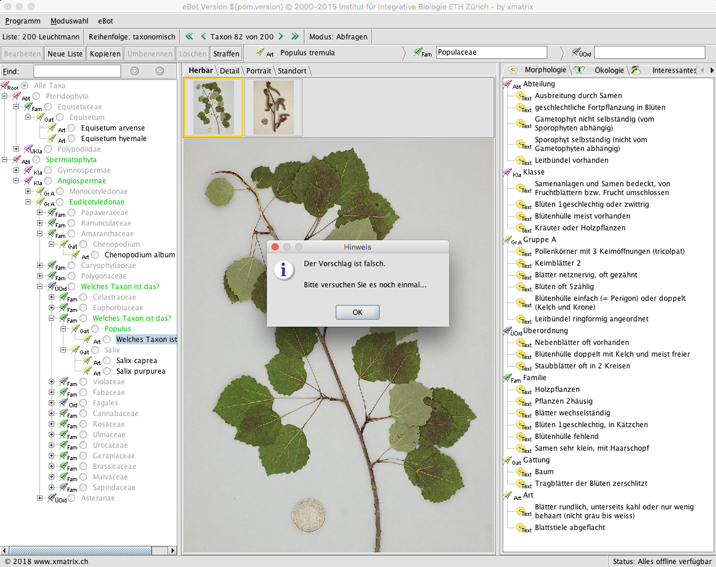
Der phylogenetische Baum kann in der Abfrage abgedeckt werden, indem der rechte Rand des Baumfensters nach links bis zum Anschlag gezogen wird.